El microbiólogo ha sido nombrado Investigador del Año por el Jurado de los Premios ABC Salud por su aportación en el desarrollo de CRISPR, la técnica del ‘corta-pega’ genético que ha supuesto toda una revolución en la medicina y la ciencia
Afable, accesible y espontáneo. La conversación con Francisco J. Martínez Mojica (Elche, 1963) es tan coloquial que a veces una olvida que está frente a quien fue candidato al Nobel. Al menos así se reflejó en las quinielas previas que los expertos y los medios de comunicación hicieron sobre los posibles premiados hasta 2020, año en el que su descubrimiento fue reconocido, aunque él no estuvo entre los elegidos. «Nunca llegué a creer en serio que tuviera alguna oportunidad», dice humilde, aunque también aliviado, porque no se prodiga mucho entre periodistas. Hoy hace una excepción para recibir el reconocimiento a Médico/Investigador del Año otorgado por el jurado de los Premios ABC Salud.
Porque, desde que gracias a unas bacterias de las salinas de Santa Pola (Alicante) descubriera el germen de una herramienta genética que ha supuesto un punto de inflexión en la ciencia que nos permite, entre otras cosas, tener trigo sin gluten, ovejas con mejor lana o tratamientos para enfermedades genéticas raras hasta ahora sin cura, se ha convertido en toda una eminencia mundial. Es algo así como el ‘padre’ de la revolución CRISPR (siglas en inglés de ‘repeticiones palindrómicas cortas agrupadas y regularmente espaciadas’), a la que Mojica bautizó sin imaginar que su potencial llegaría tan lejos.
—¿Cuándo empezó a vislumbrar que las salinas de Santa Pola guardaban aquel secreto tan revolucionario? ¿O fue algo fortuito?
—Nosotros solo pretendíamos entender cómo podían vivir en ese entorno tan extremo aquellos microorganismos. Pero, como suele pasar muchas veces en el laboratorio, buscando una cosa, te topas con otra. En este caso, nos tropezamos con secuencias repetidas en el ADN que eran extraordinarias. Eso me llamó la atención y con eso estuve diez años o más, intentando averiguar cuál era la función biológica de aquellas secuencias.
—Diez años son muchos años…
—No todo el tiempo estuvimos investigando eso. Es algo que quedó pendiente y que tuvimos la suerte de retomar a partir de 1999, momento en el que empezamos a disponer de datos suficientes como para comprobar que aquellas secuencias estaban presentes en casi todos los microorganismos de cualquier ambiente. En ese punto empezó a interesarme todavía más. Después vimos que las repeticiones eran secuencias que coincidían con virus y, finalmente, nos dimos cuenta de que esto era un sistema de inmunidad en procariotas (organismos que tienen una única célula). Durante todos esos años, realizamos diferentes experimentos, pero no teníamos ni idea de lo que estábamos haciendo. Lo único que podíamos hacer era descartar: planteábamos una hipótesis, la poníamos a prueba y comprobábamos. Así hicimos con toda clase de ideas locas.
—¿Qué hipótesis barajaron?
—Había algunas muy sesgadas. Por ejemplo, como mi trabajo de tesis estaba muy relacionado con la estructura del ADN, pensamos que era probable que estas secuencias, que se repetían una y otra vez, estuvieran afectando a la forma del ADN. De hecho, en aquella época, sobre todo a finales de los 80, se puso muy de moda la topología del ADN para intentar explicar cosas sobre las que no teníamos ninguna respuesta.
«El descubrimiento de CRISPR fue bestial. Era agosto de 2003 y vimos un ‘match’ perfecto entre una secuencia en una bacteria y otra idéntica en un virus»
—Al final, fue algo más sencillo… ¿Por qué nos costó tanto dar con un mecanismo que llevaba miles de millones de años en la naturaleza?
—Eso mismo nos decían los revisores cuando intentamos publicar el artículo -se ríe-. Que cómo era posible que nadie lo hubiera visto antes. En realidad, esto no era del todo cierto: nosotros lo vimos en el 93; pero antes, en el 87, unos científicos japoneses publicaron un artículo en el que habían secuenciado un gen (entonces, aquello era lo máximo) y observaron que habían encontrado siete repeticiones muy curiosas. Pero no siguieron con ello. Dos años más tarde publicaron otro artículo donde decían que no eran siete, sino doce secuencias, y que había otro trozo en otro sitio. Pero no sabían lo que eran y lo dejaron estar. De hecho, esos autores no volvieron a publicar nada sobre el tema.
—¿En qué momento se dan cuenta del hallazgo? ¿Cómo se sintieron?
—Fue bestial. Era agosto del 2003 y estábamos en la playa, en casa de los padres de mi mujer. Como odio la playa en verano, por el agobio y el calor, con la excusa del aire acondicionado, yo siempre me iba al laboratorio. Además, era el único momento que tenía para analizar datos. Estábamos secuenciando estas regiones repetidas en varias cepas aisladas de E. coli y vimos que entre las repeticiones había un ‘match’ perfecto: una secuencia era idéntica a otra de un virus. Y no uno cualquiera, un virus que suele infectar a E. coli. Y nosotros sabíamos que esa cepa en concreto era resistente a la infección por ese virus, pero otras cepas eran sensibles a la infección.
Entonces me puse a buscar en el ordenador microorganismos que tuvieran esas repeticiones y a subirlas a las bases de datos, a ver si había concordancias. Y empezaron a salir una detrás de otra. Miramos entonces en Streptococcus Pyogenes y de ahí extrajimos la maravillosa Cas9 que funciona como Dios. De las secuenciadas, era la única que tenía un sistema CRISPR. Fue entonces cuando entendimos que la función de cumplían estas secuencias era de inmunidad y que, de alguna forma, constituían unos almacenes de memoria genéticos donde reconocían, recordaban y reconocían infecciones que habían afectado en un pasado a sus ancestros.
—¿Imaginaba en aquel entonces hasta dónde llegarían las aplicaciones?
—No. De hecho, cuando mandamos el artículo, no coló. Y eso que nosotros pensábamos que iba a tener una gran repercusión a nivel clínico, biotecnológico e incluso ambiental. Porque tener un sistema programable para hacer que una bacteria sea susceptible o resistente a un virus iba a tener un impacto enorme. Por ejemplo, con los lácteos: tú añades fermentos y a veces aquello no va para delante. Esto es porque ha habido un virus y lo ha contaminado todo. Entonces, con CRISPR, puedes blindar a estas bacterias para que sean resistentes. Pero yo trabajo con microorganismos, y tengo una mente de microbiólogo que no va más allá. Y no pensé en todo lo que se podía hacer con aquello.
«Ganar el Nobel me lo planteé como algo bueno para el país, pero que podía ser un cambio muy gordo para mí»
—¿Y cuando Emmanuelle Charpentier y Jennifer A. Doudna encontraron la forma de aplicarlo a cualquier célula?
—Cuando publicaron el artículo en 2012, vino un colaborador a decírmelo. Que con aquello se podía hacer edición genética. Yo le dije ‘¿y a mí qué me cuentas?’. Nos olvidamos del tema hasta que unos meses más tarde aparecieron dos artículos más diciendo que habían editado el genoma de células humanas con una eficiencia increíble utilizando CRISPR. Ahí vi que la cosa tenía muy buena pinta. Después fue una explosión de artículos científicos: todo el mundo estaba haciendo edición CRISPR. Y ahora mismo todo el mundo se ha olvidado prácticamente de que existen otras alternativas de edición genética.
—De hecho, hay una polémica reglamentación de la UE que equipara las nuevas formas de edición genética con transgénicos, pero que todo el mundo relaciona con CRISPR.
—Efectivamente. Bueno, a la gente le salen sarpullidos cuando oye el término ‘transgénico’. En este caso se trata de un organismo al que le han añadido ADN de otro. Sin embargo, con CRISPR no queda ningún material genético de otro ser vivo dentro del organismo modificado. La herramienta solo hace su trabajo: le puedes añadir la proteína, le puedes añadir la guía, le puedes añadir los mensajeros para que se produzcan esas proteínas, pero luego te lo quitas de encima. O sea, no se queda ahí, en esa célula. De manera que puedes hacer los mismos cambios que ocurren en la naturaleza aleatoriamente, pero dirigidos.
—Es una herramienta que ha ganado un Nobel. Sin embargo, usted no estuvo entre los premiados aunque estaba en las quinielas de mucha gente, incluidos colegas suyos…
—Yo en su momento me lo planteé como algo bueno para el país, pero que podía ser un cambio demasiado gordo en mi vida. Sin embargo, nunca llegué a creerme de verdad que tuviera ninguna posibilidad.
—Pero mucha gente se enfadó cuando usted no fue uno de los elegidos.
-Lo sé, lo sé. Pero estaba claro que lo iban a dar por la tecnología, no al descubridor de un sistema inmunológico de los procariotas. Solo en una ocasión, con Werner Arber, el descubridor de los sistemas de enzimas de restricción, se le incluyó en el ‘paquete’. Y algunos pensaron que esto podía ser equivalente. Pero aquí estaba claro que había dos personas que tenían que estar. Quedaba un tercer sitio pero, evidentemente, no lo tuvieron claro.

—¿Cómo fue ese momento en el que se entera de que les han dado el Nobel a Charpentier y a Doudna?
—Estaba en mi despacho y vino un periodista a decirme que iban a dar los Nobel, que qué hacía que no lo veía. Lo seguimos desde su móvil. Y entonces hablan de que se lo dan a la edición genética y luego dicen el nombre de Jennifer y Emmanuel. Cinco minutos más tarde, aparece un colaborador en el laboratorio. ‘¡Que no se lo han dado a Siksnys!’ [Virginijus Siksnys descubrió casi de forma paralela la herramienta, si bien por problemas con la publicación, se adelantaron Charpentier y Doudna]. El periodista se quedó de plástico con aquel comentario.
—Esta herramienta nos permite cortar, pegar, activar, desactivar genes… incluso hacer que el ADN ‘evolucione’ dentro de la célula. ¿Hay techo para esta técnica o seguimos sin verlo?
—No tiene tope. Es absolutamente increíble lo que ha pasado desde el año 2013 hasta ahora. Yo ya he perdido la cuenta de los artículos que hay. Estamos constantemente descubriendo variantes que hacen cosas distintas. Hasta a mí me cuesta encontrar los artículos que realmente me interesan para mi trabajo, porque todo el mundo está haciendo edición genética. Y no solo eso, también se están utilizando como antimicrobianos, como sistemas de diagnóstico molecular para detectar virus como el del COVID, de ADN, de ARN…
—Y tratamientos genéticos como el que acaban de aprobar Reino Unido y EE.UU. para la enfermedad de células falciformes y talasemia.
—Y Europa vendrá después. [Unos días después de la entrevista, la Agencia Europea de Medicamentos (EMA) recomendaba la aprobación de este medicamento].
El primer tratamiento con edición genética supera su primer examen: «Es una esperanza para muchas enfermedades genéticas»
Un panel de expertos de la FDA cree que la terapia es segura para tratar un tipo de anemia potencialmente mortal
La agencia del medicamento de Estados Unidos, aún tiene que dar la luz verde definitiva. Podría costar millones de euros por paciente
CRISPR, la revolución genética que ya nos podemos comer
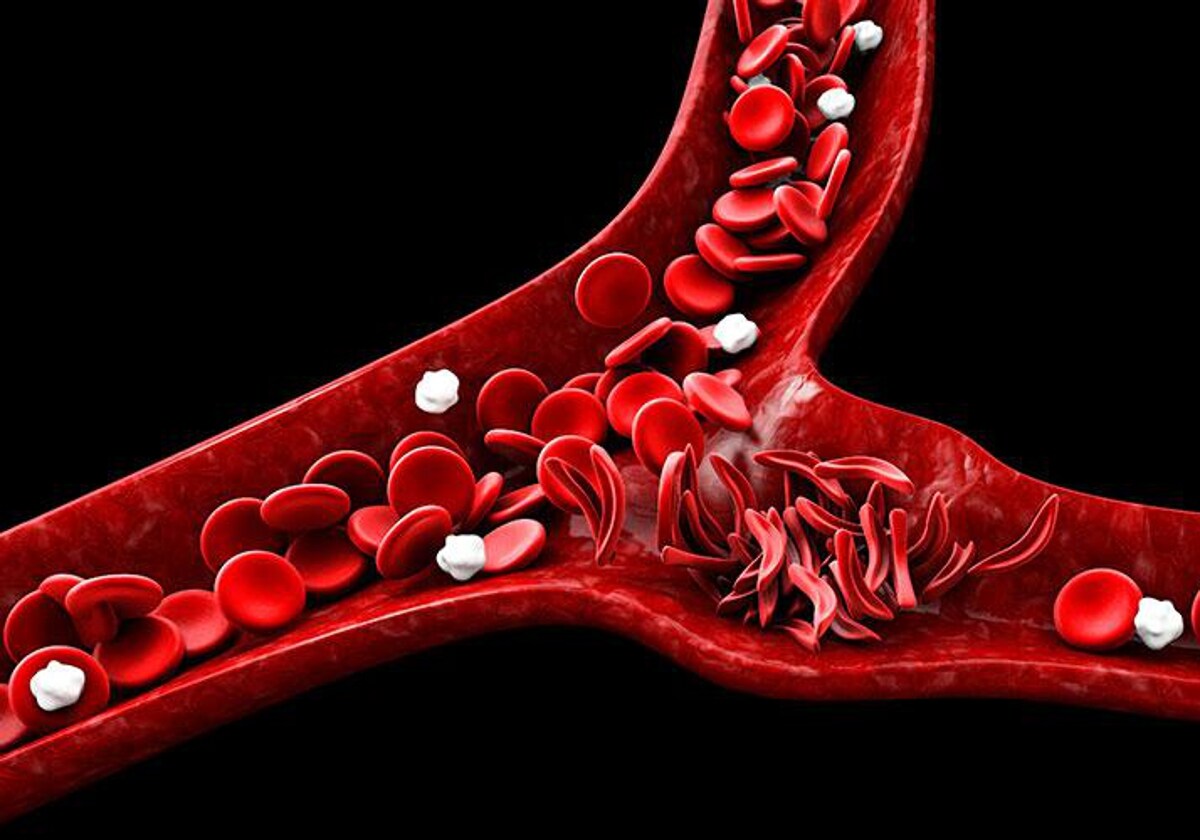
La anemia falciforme está causada por una mutación genética que fabrica glóbulos rojos anómalos. Deformes y pegajosos pueden formar grumos que bloqueen los vasos sanguíneos. Causan ictus, daño orgánico y crisis de dolor
El primer tratamiento basado en la edición genética del ser humano ha pasado ya el primer filtro para su aprobación. El panel de expertos que asesora a la FDA, la Agencia del Medicamento de Estados Unidos, ha concluido que el fármaco no solo es eficaz, sino seguro. La última palabra para su aprobación definitiva la tiene este organismo federal que deberá tomar la decisión definitiva antes del 8 de diciembre.
Se evalúa su uso para tratar la anemia falciforme, una rara enfermedad sanguínea muy dolorosa para la que no existe un tratamiento eficaz. Pero sería también el comienzo de una revolución médica, el disparo de salida para una tecnología que podría revolucionar la forma de tratar las enfermedades.
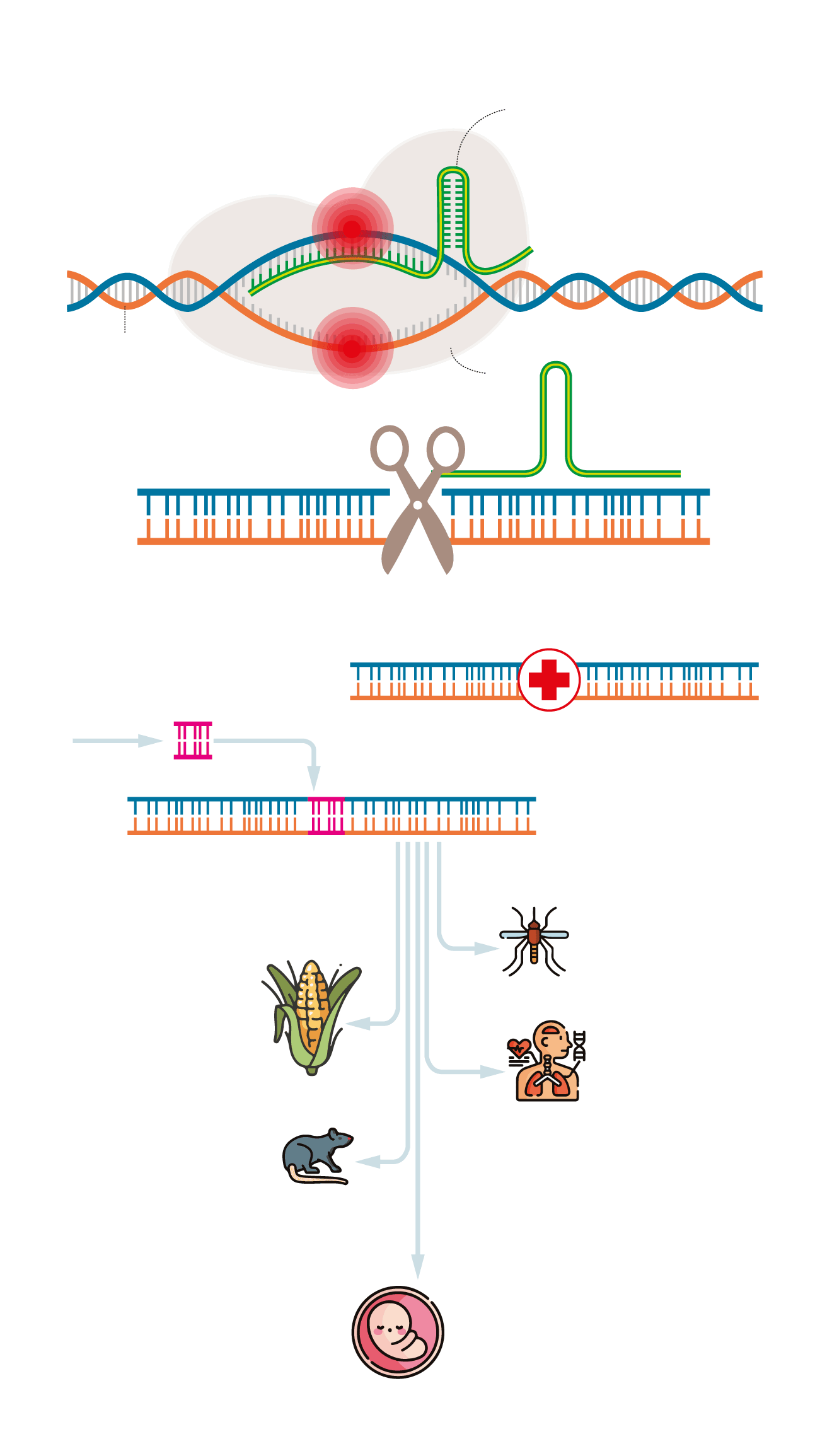
La protagonista de esa revolución es una herramienta de nombre casi impronunciable: CRISPR/Cas9 (léase crísper) que permite editar el genoma humano, quitando y poniendo genes a voluntad. De ahí que se bautizara como ‘corta pega’ genético. La herramienta se utiliza ya en agricultura para conseguir cultivos mejorados. En medicina, se ha recurrido a ella de forma experimental con dos centenares de enfermos de diferentes patologías. La autorización de la FDA supondría el salto a la clínica y a un uso más generalizado de los pacientes.
«Lo que suceda con esta terapia marcará la pauta para otros tratamientos CRISPR que ya están siendo testados. Hay cerca de 90 ensayos clínicos registrados en diversas fases de análisis) y que pueden traer la esperanza terapéutica a muchas personas que conviven con enfermedades congénitas, la mayoría consideradas raras», pronostica Lluis Montoliu, investigador del CSIC.
Adiós a las crisis de dolor
El tratamiento que evalúa Estados Unidos se llama exa-cel y ha sido desarrollado conjuntamente por Vertex Pharmaceuticals de Boston y la compañía suiza CRISPR Therapeutics. En los ensayos clínicos presentados se muestra cómo los pacientes con anemia falciforme tratados se liberan de los efectos debilitantes y dolorosos de esta enfermedad crónica y mortal.
La vida no es fácil para estos enfermos. Sus glóbulos rojos, que deberían ser circulares, adoptan una forma de media luna y se convierten en pegajosos y rígidos lo que hace que se aglutinen. Estos ‘grumos‘ ocluyen los vasos sanguíneos y privan a los tejidos de oxígeno, produciendo daños graves en los órganos y episodios de dolor agudo. Quienes lo sufren se ven obligados a someterse a entre 15 y 30 transfusiones al año y soportan crisis de dolor repentinas y muy severas.
El nuevo tratamiento elimina casi por completo los síntomas. Lo hace favoreciendo la producción de una forma de hemoglobina (la proteína que lleva oxígeno desde los pulmones al resto del cuerpo) que normalmente sólo se fabrica en los fetos en desarrollo. Así se obtiene un poco de hemoglobina que no está deformada y se amortiguan los efectos de la forma anormal.
Posibles errores
Vertex y CRISPR Therapeutics informaron de que, a los nueve meses del tratamiento, 31 de los 32 participantes en su ensayo clínico no habían sufrido ni una sola crisis de dolor. Antes del tratamiento, habían tenido una media de unas cuatro cada año.
Para aplicar el tratamiento, los médicos extraen primero células madre productoras de sangre de enfermos. A continuación, las células se editan genéticamente para cortar el ADN y una molécula que guía a la enzima hasta un tramo de ADN diana en el gen BCL11A.
Una vez en esa región, la enzima Cas9 corta ambas cadenas de ADN. A continuación, los mecanismos naturales de reparación del ADN de la célula vuelven a unir las hebras. Pero esos mecanismos pueden equivocarse, lo que significa que a menudo introducen errores en la secuencia de ADN.
Beneficios y riesgos
Esta es una complicación no esperada pero posible con las herramientas CRISPR. De ahí que sea tan importante el veredicto de este panel de expertos sobre su seguridad. «De momento, en el poco tiempo que llevan los relativamente pocos pacientes tratados en ensayos clínicos, no han aparecido problemas significativos, pero es posible que la agencia reguladora norteamericana requiera más estudios para garantizar la seguridad del tratamiento», argumenta el investigador del CSIC.
Existe también preocupación por la posibilidad de que este tipo de terapias eleven el riesgo de cáncer. Algunos estudios han demostrado que las personas con anemia falciforme podrían ser más propensas a padecer cánceres hematológicos. Si entre las células extirpadas, alteradas y reinfundidas había células precancerosas, el tratamiento podría favorecer su desarrollo. Es una complicación, aún por estudiar que se conocerá a medio plazo.
En contra, también está su elevado precio. Se espera que el nuevo fármaco cueste millones de euros por paciente, aunque el coste a largo plazo de los enfermos ahora es también muy elevado.
Otras alternativas con terapia génica
La estrategia de Vertex no es la única en el tratamiento de esta anemia tan grave. La FDA valorará también en diciembre otra posible cura, aunque en esta ocasión con una terapia génica desarrollada por la empresa estadounidense Bluebird Bio. «Se trata de una terapia más tradicional, no basada en CRISPR, que reintroduce una copia correcta del gen afectado en las células de los pacientes», explica Montoliu.










